制药用水及受控环境中寡营养微生物的生物负载监测
时间:
摘要:目的 筛选适用于制药环境中寡营养微生物的监测方法,明确制药用水及洁净环境等寡营养条件下的生物负载及微生物特征。方法 比较R₂A和TSA培养基对实验室纯水中微生物在不同温度、培养基及培养时间的计数结果,优化寡营养条件下微生物的培养条件和检测方法;采用传统方法与寡营养检测方法同步监测制药生产环境中寡营养微生物,结合16S rDNA测序和MALDI-TOF MS技术,对分离的微生物开展多相微生物鉴定及分析。结果 菌落计数方法、培养基种类和培养时间对微生物计数有显著性影响;寡营养R₂A培养基分离环境微生物的效果优于TSA培养基,两种培养基的微生物分离谱差异较大,R₂A培养基中革兰阴性菌的分离率达37.0%,而TSA培养基中仅为14.0%;此外,初次分离于R₂A培养基的多株条件致病微生物在TSA培养基中生长缓慢,易被传统监测方案漏检。结论 随着制药工艺发展,人源性革兰阳性球菌污染比例将逐步下降,选用寡营养微生物监测方法作为传统技术的补充,有助于早期发现生产环境中潜在的不可接受微生物污染风险。
关键词:寡营养微生物;制药用水;受控环境;生物负载;不可接受微生物
论文《制药用水及受控环境中寡营养微生物的生物负载监测》发表在《药物分析杂志》,版权归《药物分析杂志》所有。本文来自网络平台,仅供参考。
Abstract:Objective To screen suitable monitoring methods for oligotrophs and clarify the bioburden and microbial characteristics of oligotrophic environments such as pharmaceutical water systems and cleanrooms. Methods The culture conditions and detection methods for microorganisms under oligotrophic conditions were optimized by comparing the counting results of microorganisms in laboratory pure water using R₂A and TSA media at different temperatures and incubation times. Meanwhile, oligotrophs in pharmaceutical production environments were monitored simultaneously by traditional and oligotrophic detection methods. Combined with 16S rDNA sequencing and MALDI-TOF MS technologies, multiphasic microbial identification and analysis were conducted on the isolated microorganisms. Results Colony counting methods, medium types, and incubation times significantly affected microbial enumeration. The oligotrophic R₂A medium was more effective in isolating environmental microorganisms than TSA medium, with distinct microbial isolation profiles between the two media. The isolation rate of Gram-negative bacteria in R₂A medium reached 37.0%, while that in TSA medium was only 14.0%. In addition, several strains of opportunistic pathogens initially isolated from R₂A medium grew slowly in TSA medium and were easily missed by traditional monitoring methods. Conclusion With the development of pharmaceutical processes, the proportion of human-derived Gram-positive cocci contamination will gradually decrease. Timely use of oligotrophic microbial monitoring methods as a supplement to traditional technologies helps detect potential objectionable microorganism contamination risks in the production environment at an early stage.
Keywords:oligotrophs; pharmaceutical water; controlled environment; bioburden; objectionable microorganism
1 引言
寡营养微生物(oligotrophs)是一类可在有机物含量极低环境(含碳量1~15mg·L⁻¹)中存活并保持一定生长速率的微生物[1],主要由非发酵革兰阴性菌组成,广泛存在于天然水体、空气和土壤等自然环境中[2]。这类微生物耗氧量低、营养需求少,极易在药品生产环境及其水系统中长期存活和生长[3],且具有较强的环境耐受性,可通过产生生物被膜、生物毒素等自我保护机制抵抗消毒剂,同时携带多种抗生素耐药性,还能通过质粒在微生物群落间传播耐药性和抗逆性[4-9]。
在营养选择压力下,寡营养微生物常处于活的不可培养状态(Viable but Non-Culturable, VBNC),难以通过常规手段分离培养[10]。已知的寡营养微生物主要包括不动杆菌属(Acinetobacter spp.)、甲基杆菌属(Methylobacterium spp.)、假单胞菌属(Pseudomonas spp.)等[11],其中不少为条件致病菌,每年可引起上万人感染[12-13],还可能随药品原辅料和生产工艺进入终产品,导致药品污染,成为内毒素的主要来源之一,对易感人群、免疫抑制和免疫缺陷患者的安全构成巨大威胁[9,14-15]。
目前制药生产过程中生物负载监控主要采用胰酪大豆胨琼脂(TSA)、沙氏葡萄糖琼脂(SDA)等富营养培养基,适合培养嗜中温微生物,这类微生物以人源革兰阳性球菌为主要污染源[10,16-19]。但寡营养微生物可能处于VBNC状态,生长缓慢,首次复苏时难以适应富营养条件,易被其他微生物群落掩盖,难以在日常监测中快速形成可观察的菌落[20-22],因此传统富营养培养方法并不适用于寡营养微生物监测。
随着基因治疗产品等新兴生物制品进入全封闭、自动化生产新阶段,操作人员直接接触产品的概率降低,人源性微生物污染预期将显著减少,而物料、空气和制药用水等带来的寡营养微生物可能成为未来药品污染的重要因素[15,23]。探索寡营养微生物的有效监测方法迫在眉睫。常见的寡营养培养基主要有R₂A培养基和低浓度SCDA培养基等[24-25],国际主流药典已普遍选用R₂A培养基作为制药用水微生物监测的首选培养基[26-27]。本研究通过筛选适宜的寡营养微生物培养方法,监测制药用水、洁净空气和洁净表面等典型环境,解决传统监测技术漏检问题,结合多相鉴定技术分析微生物种类与群落分布,为企业建立全面的环境微生物监测方案提供参考。
2 实验材料、仪器与试剂
2.1 实验材料
培养基:R₂A培养基和TSA培养基采购自北京陆桥技术有限责任公司,按厂家说明书制备;表面菌采样平皿规格为55mm,其余平皿规格为90mm。
样本:纯化水样本来自上海市食品药品检验研究院微生物实验室管道纯化水;环境样本来自国内某生物制药公司细胞生产车间。
2.2 实验仪器
Biotyper基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS,布鲁克公司)、Veritii PCR扩增仪(赛默飞公司)、Quantity One紫外凝胶成像仪(伯乐公司);数据统计软件为OriginPro 2018。
2.3 实验试剂
细菌基因组DNA提取试剂盒(北京天根生化科技公司);细菌核酸扩增及测序引物:上游引物27F(5’-AGAGTTTGATCCTGGCTCAG-3’)、下游引物1492R(5’-ACGGCTACCTTGTTACGACTT-3’);DNA引物合成与测序服务由生工生物工程(上海)股份有限公司提供。
3 实验方法
3.1 药品检测实验室纯水系统微生物监测
采用直接接种法和薄膜过滤法(滤膜孔径≥0.45μm,直径约50mm)监测10个管道纯水使用点,每个使用点采样量不少于100mL。
直接接种法:取4份纯化水各1mL,分别接种于TSA培养基和R₂A培养基,每种培养基各2个平皿,分别置于20~25℃和30~35℃培养。
薄膜过滤法:取4份纯化水各10mL,过滤后接种于两种培养基,培养条件同上。
记录培养第3、5、7d时的需氧菌总数计数结果,计算菌落数平均值(CFU·mL⁻¹),分析培养基、培养时间、温度和接种方法对计数结果的影响。
3.2 制药企业环境微生物监测
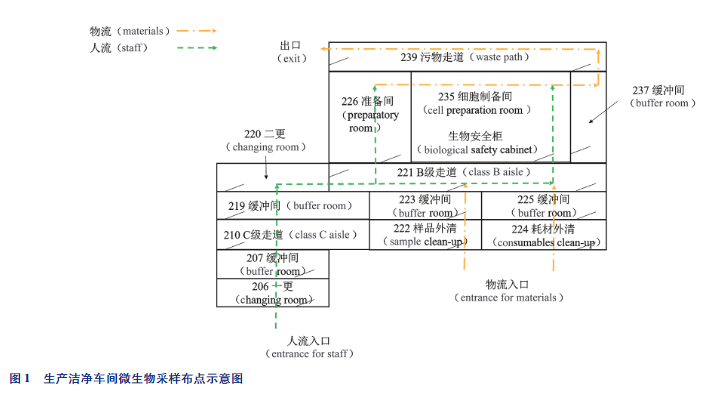
按2020年版《中华人民共和国药典》通则9205要求对制药洁净室采样,采样布点示意图见图1。采用R₂A和TSA培养基分别收集沉降菌、浮游菌、设备与操作人员表面菌,采样信息见表1:TSA培养基监测的采样点取2个样品,R₂A培养基监测的采样点取1个样品,墙面均仅取1个样品。采样后置30~35℃培养7d,统计需氧菌总数计数结果。
图1 生产洁净车间微生物采样布点示意图
(注:物流(materials)、人流(staff)、出口(exit)、污物走道(waste path)、准备间(preparatory room)、细胞制备间(cell preparation room)、缓冲间(buffer room)、二更(2nd changing room)、B级走道(class B aisle)、C级走道(class C aisle)、一更(1st changing room)、生物安全柜(biological safety cabinet)、样品外清(sample clean-up)、耗材外清(consumables clean-up)、物流入口(entrance for materials)、人流入口(entrance for staff))
表1 生产洁净车间微生物采样点信息汇总
| 房间 | 功能 | 洁净度 | R₂A培养基采样点数 | TSA培养基采样点数 |
| --| --| --| --| --|
| | | | AB(浮游菌) | ST(沉降菌) | SF(表面菌) | AB(浮游菌) | ST(沉降菌) | SF(表面菌) |
| 206 | 一更(1st changing room) | D | 2 | 2 | 1 | | | |
| 207 | 缓冲间(buffer room) | C | 2 | 2 | | 1 | 1 | |
| 210 | C级走道(class C aisle) | C | | | 墙面(wall):2 | | | 墙面(wall):1 |
| 219 | 缓冲间(buffer room) | C | | | 门把手(door handle):2 | | | 内侧把手(inside door handle):1 |
| 220 | 二更(2nd changing room) | B | | | 门把手(door handle):2、墙面(wall):2、台面(desktop):1 | | | 内侧把手(inside door handle):1、墙面(wall):1 |
| 226 | 准备间(preparatory room) | B | | | 门把手(door handle):1、墙面(wall):2、冰箱(refrigerator):1、台面(desktop):1 | | | 冰箱(refrigerator):1、台面(desktop):1 |
| 221 | B级走道(class B aisle) | B | | 1 | 门把手(door handle):2、墙面(wall):2 | 1 | | 内侧把手(inside door handle):1、墙面(wall):1 |
| 223 | 缓冲间(buffer room) | B | | | 门把手(door handle):2、墙面(wall):2 | | | |
| 225 | 缓冲间(buffer room) | B | | | 门把手(door handle):2、墙面(wall):2 | | | |
| 235 | 细胞制备间(cell preparation room) | B | 1 | | 门把手(door handle):2、墙面(wall):2、台面(desktop):1、设备表面(equipment surface):7 | | 1 | 内侧把手(inside door handle):2、墙面(wall):2、台面(desktop):1、设备表面(equipment surface):2 |
| 235 | 生物安全柜(biological safety cabinet) | A | 2 | 2 | 设备表面(equipment surface):4 | 2 | 2 | 设备表面(equipment surface):2 |
| 237 | 缓冲间(buffer room) | B | | | 门把手(door handle):2、墙面(wall):2 | | | |
| 239 | 污物走道(waste path) | C | 1 | | 墙面(wall):2 | 1 | | 墙面(wall):1 |
| 采样人员(sampler) | | | | | 手套(gloves):2 | | | 手套(gloves):2 |
| 总计(total) | | | 9 | 10 | 52 | 6 | 6 | 21 |
(注:a. AB:浮游菌(airborne microbes);ST:沉降菌(settling microbes);SF:表面菌(surface microbes);b. “-”表示未设置监测点)
3.3 微生物的鉴定
挑取每个监测平板中形态不同的微生物,分别划线接种至R₂A和TSA培养基,置30~35℃培养纯化。采用MALDI-TOF MS技术鉴定,可信度小于2.0的微生物采用16S rDNA测序技术进一步鉴定。
MALDI-TOF MS鉴定[28]:采用原位甲酸提取法,将微生物均匀涂抹于检测靶板,自然干燥后加入1μL 70%甲酸溶液,干燥后加入1μL α-氰基-4-羟基肉桂酸(HCCA)基质溶液,晾干后上机检测。
16S rDNA测序鉴定[29]:将微生物用500μL生理盐水分散,按试剂盒说明书提取核酸,置100μL TE缓冲液-20℃保存。PCR反应体系(50μL):DNA模板2μL、rTag DNA聚合酶1U、1×PCR缓冲液、4种dNTP各200μmol·L⁻¹、上下游引物各200pmol·L⁻¹,加纯化水补足。PCR反应条件:95℃变性1min;95℃变性30s、56℃退火30s、72℃延伸90s,共30个循环;72℃延伸10min,产物置-20℃保存测序。测序数据拼接后与数据库比对确定微生物种类。
4 实验结果
4.1 纯化水系统微生物监测结果
不同培养条件对纯化水系统生物负载计数结果的影响见图2。菌落计数方法、培养基种类和培养时间对微生物计数有显著性影响:R₂A培养基的微生物分离效果优于TSA培养基;直接接种法的菌落计数结果优于薄膜过滤法,可能因薄膜有效过滤面积小,菌落易重叠且微小菌落不易观察;30~35℃培养条件下的计数结果整体高于20~25℃;随着培养时间延长,菌落数逐渐增加,7d时计数结果最稳定。
图2 不同影响因素下纯化水系统生物负载计数结果
(注:A. 以直接接种法与薄膜过滤法为分类因素;B. 以R₂A培养基和TSA培养基为分类因素;C. 以培养时间3、5、7d为分类因素;D. 以培养温度20~25℃和30~35℃为分类因素)
在10个纯化水采样点,R₂A和TSA培养基共分离鉴定出68株微生物:R₂A培养基分离菌株占42.6%(29/68),TSA培养基占57.4%(39/68)。两种培养基监测到的微生物种群差异较大(表3),R₂A培养基中分离出3种在TSA培养基上不易分离或生长的条件致病菌株,即高地芽孢杆菌(Bacillus altitudinis)、土壤短芽孢杆菌(Brevibacillus agri)和桥石短芽孢杆菌(Brevibacillus choshinensis);皮氏罗尔斯顿菌(Ralstonia pickettii)是报道对水基质产品有潜在危害的不可接受微生物,仅在R₂A培养基中分离得到。
表3 TSA与R₂A培养基分离微生物汇总表
| 初次分离的培养基 | 菌株 | 特性 |
| --| --| --|
| 初次仅分离于TSA培养基 | 米兰农霉菌(Agromyces mediolanus) | 真菌(fungi) |
| | 巨大芽孢杆菌(Bacillus meqaterium) | 产芽孢(spore producing) |
| | 壁芽孢杆菌(Bacillus muralis) | 产芽孢(spore producing) |
| | 短短芽孢杆菌(Brevibacillus brevis) | 产芽孢(spore producing) |
| | 滕黄短小杆菌(Curtobacterium luteum) | 条件致病菌(opportunistic pathogen) |
| | 埃吉类芽孢杆菌(Paenibacillus elqii) | |
| | 解葡聚糖类芽胞杆菌(Paenibacillus glycanilyticus) | |
| | 山羊葡萄球菌(Staphylococcus caprae) | 条件致病菌(opportunistic pathogen) |
| | 嗜麦芽窄食单胞菌(Stenotrophomonas maltophilia) | 条件致病菌(opportunistic pathogen) |
| 初次仅分离于R₂A培养基 | 高地芽孢杆菌(Bacillus altitudinis) | 产芽孢(spore producing) |
| | 土壤短芽孢杆菌(Brevibacillus agri) | 产芽孢(spore producing) |
| | 桥石短芽孢杆菌(Brevibacillus choshinensis) | 产芽孢(spore producing) |
| TSA和R₂A共同分离的菌株 | 蜡样芽孢杆菌(Bacillus cereus) | 产芽孢(spore producing) |
| | 海泥芽孢杆菌(Bacillus oceanisediminis) | 产芽孢(spore producing) |
| | 简单芽孢杆菌(Bacillus simplex) | 产芽孢(spore producing) |
| | 类短短芽孢杆菌(Brevibacillus parabrevis) | 产芽孢(spore producing) |
| | 罗伊氏短芽孢杆菌(Brevibacillus reuszeri) | 产芽孢(spore producing) |
| | 腐殖质类芽孢杆菌(Paenibacillus humicus) | |
| | 皮氏罗尔斯顿菌(Ralstonia pickettii) | 条件致病菌(opportunistic pathogen) |
4.2 制药环境寡营养微生物监测结果
对制药生产车间104个监测数据统计分析(排除同一采样点两种培养基均为零值的结果),R₂A和TSA培养基的监测结果均满足2020年版《中华人民共和国药典》四部通则9205对洁净环境的要求。两种培养基共分离微生物56株,其中R₂A培养基分离菌株占46.7%,TSA培养基占53.3%。
微生物分离率分析显示(表5、图3):R₂A培养基中革兰阴性菌的分离率为37.0%,革兰阳性菌为63.0%;而TSA培养基中革兰阴性菌分离率仅14.0%,革兰阳性菌达86.0%。以“属”分类,R₂A培养基更易分离环境中革兰阴性微生物,如寡养单胞菌属(Stenotrophomonas spp.)、莫拉菌属(Moraxella spp.)、假单胞菌属(Pseudomonas spp.)等;TSA培养基更适合监测人源性污染微生物,葡萄球菌属(Staphylococcus spp.)分离率高达39.3%。此外,初次分离于R₂A培养基的多株条件致病菌在TSA培养基中生长缓慢,培养超过5d仍难以识别,易被传统监测漏检。
表5 制药环境R₂A和TSA培养基中典型微生物的分离率
| 序号 | 微生物种类 | 革兰染色 | 分离率/% | |
| --| --| --| --| --|
| | | | R₂A培养基 | TSA培养基 |
| 1 | 微球菌属(Micrococcus spp.) | G+ | 22.5 | 19.6 |
| 2 | 芽孢杆菌属(Bacillus spp.) | G+ | 14.3 | 8.9 |
| 3 | 寡养单胞菌属(Stenotrophomonas spp.) | G| 14.3 | 1.8 |
| 4 | 葡萄球菌属(Staphylococcus spp.) | G+ | 12.2 | 39.3 |
| 5 | 微小杆菌属某些种(Microbacterium spp.) | G+ | 8.2 | 7.1 |
| 6 | 莫拉菌属某些种(Moraxella spp.) | G| 6.1 | 1.8 |
| 7 | 假单胞菌属(Pseudomonas spp.) | G| 4.1 | 1.8 |
| 8 | 分枝盐场单胞菌属(Salinarimonas spp.) | G| 4.1 | 0.0 |
| 9 | 短杆菌属(Brachybacterium spp.) | G+ | 2.0 | 0.0 |
| 10 | 短杆菌属某些种(Brevibacterium spp.) | G+ | 2.0 | 0.0 |
| 11 | 戈登氏菌属(Gordonia spp.) | G+ | 2.0 | 1.8 |
| 12 | 甲基杆菌属(Methylobacterium spp.) | G| 2.0 | 0.0 |
| 13 | 副球菌属(Paracoccus spp.) | G| 2.0 | 0.0 |
| 14 | 鞘氨醇杆菌属(Sphingobacterium spp.) | G| 0.0 | 3.6 |
| 15 | 不动杆菌属(Acinetobacter spp.) | G| 0.0 | 1.8 |
| 16 | 嗜碱杆菌属(Alkalihalobacillus spp.) | G+ | 0.0 | 3.6 |
| 17 | 布鲁氏菌属(Brevundimonas spp.) | G| 0.0 | 1.8 |
| 18 | 皮肤球菌属(Dermacoccus spp.) | G+ | 0.0 | 1.8 |
| 19 | 库克氏菌属(Kocuria spp.) | G+ | 0.0 | 1.8 |
| 20 | 瑙曼氏菌属(Naumannella spp.) | G+ | 0.0 | 1.8 |
| 21 | 泛菌属某些种(Pantoea spp.) | G| 0.0 | 1.8 |
图3 制药环境中R₂A和TSA培养基中微生物的分离率
(注:A. R₂A培养基;B. TSA培养基)
5 讨论
本研究比较了R₂A和TSA培养基在制药用水系统和洁净环境生物负载监测中的应用效果,结果表明寡营养R₂A培养基在分离环境微生物,尤其是革兰阴性菌方面具有显著优势,其分离率(37.0%)远高于TSA培养基(14.0%)。这是因为R₂A培养基营养成分浓度低,更符合寡营养微生物的生长需求,且在7d的充分培养时间内,微生物形成独立菌落前不易互相掩盖,有利于非优势种群的发现和分离[26]。
传统TSA培养基适合监测常见人源性污染微生物(主要为革兰阳性菌),且检测时间较短(2~3d),有利于控制时间成本;而R₂A培养基更适合监测环境来源的寡营养微生物,可复苏更丰富的微生物种群,弥补传统方法的漏检风险。随着制药工艺的进步,尤其是细胞治疗、基因治疗等新兴生物制药中隔离系统和密闭式生产工艺的广泛应用,人员与生产过程的直接接触大幅减少,人源性微生物污染比例将显著下降,而自然界中广泛存在的寡营养微生物(如洋葱伯克霍尔德菌群、罗尔斯通菌属、寡氧单胞菌属等)可能成为药品工艺污染监控的重点[9,12-13]。
此外,本研究发现薄膜过滤法的计数效果不如直接接种法,可能与薄膜过滤过程中微生物吸附损失、菌落重叠等因素有关,因此在制药用水寡营养微生物监测中,直接接种法更具优势。同时,部分寡营养条件致病菌在TSA培养基中生长缓慢,易被传统监测方案漏检,进一步说明引入寡营养监测技术的必要性。
企业在建立环境微生物监测方案时,可以传统监测技术为基础,辅以寡营养检测技术,基于风险评估原则构建质量控制体系。通过多相鉴定技术分析微生物污染风险,指导环境消毒和方法验证等过程控制,消除潜在隐患。本研究初步形成了完善的寡养环境微生物过程监控模式,为制定微生物污染控制策略提供了有效信息,有助于完善制药环境微生物质量监控体系,为企业质量风险管理提供案例参考。
未来可进一步优化寡营养微生物的培养条件和检测方法,扩大样本覆盖范围,深入研究寡营养微生物的污染溯源及传播机制,为制药行业微生物污染控制提供更全面的技术支撑。
参考文献
[1] KUZNETSOV SI, DUBININA GA, LAPTEVANA. Biology of oligotrophic bacteria[J]. Annu Rev Microbiol, 1979, 33(1): 377.
[2] WILLIAMS MM, ARMBRUSTER CR, ARDUINO MJ. Plumbing of hospital premises is a reservoir for opportunistically pathogenic microorganisms: a review[J]. Biofouling, 2013, 29(2): 147.
[3] FAVERO MS, CARSON LA, BOND WW, et al. Pseudomonas aeruginosa: growth in distilled water from hospitals[J]. Science, 1971, 173(3999): 836.
[4] FEAZELL M, BAUMGARTNER LK, PETERSON KL, et al. Opportunistic pathogens enriched in showerhead biofilms[J]. Proc Natl Acad Sci USA, 2009, 106(38): 16393.
[5] GAO XL, SHAO MF, WANG Q, et al. Airborne microbial communities in the atmospheric environment of urban hospitals in China[J]. J Hazard Mater, 2018, 349(5): 10.
[6] BEERD D, SRINIVASAN R, STEWART PS. Direct measurement of chlorine penetration into biofilms during disinfection[J]. Appl Environ Microbiol, 1994, 60(12): 4339.
[7] 韩东东, 郝振宇, 高广海, 等. 寡营养细菌及其生态作用和应用的研究进展[J]. 微生物学通报, 2012, 39(4): 526.
[8] ZLATKIN IV, VISHNEVETSKAIA OI, NIKITIN DI. Some aspects of antibiotic resistance of oligotrophic bacteria[J]. Antibiot Chemother, 1991, 36(3): 34.
[9] MITTELMAN MW, JONES ADG. A pure life: the microbial ecology of high purity industrial waters[J]. Microb Ecol, 2018, 76(1): 9.
[10] RAMAMURTHY T, GHOSH A, PAZHANI GP, et al. Current perspectives on viable but non-culturable (VBNC) pathogenic bacteria[J]. Front Public Health, 2014, 2(103): 1.
[11] K?KIZ I, GR?BNER K, BOHUS V, et al. Application of special oligotrophic media for cultivation of bacterial communities originated from ultrapure water[J]. Acta Microbiol Immunol Hung, 2013, 60(3): 345.
[12] FALKINHAM JO, PRUDEN A, EDWARDS M. Opportunistic premise plumbing pathogens: increasingly important pathogens in drinking water[J]. Pathogens, 2015(4): 373.
[13] KOVALEVA J, DEGENER JE, VAN DER MEI HC. Methylobacterium and its role in healthcare-associated infection[J]. J Clin Microbiol, 2014, 52(5): 1317.
[14] TADA Y, IHMORI M, YAMAGUCHI J. Oligotrophic bacteria isolated from clinical materials[J]. J Clin Microbiol, 1995, 33(2): 493.
[15] NAGARKAR PP, RAVETKAR SD, WATVEM G. Oligophilic bacteria as tools to monitor aseptic pharmaceutical production units[J]. Appl Environ Microbiol, 2001, 67(3): 1371.
[16] FUHRMAN JA, CAMPBELL L. Marine ecology: microbial microdiversity[J]. Nature, 1998, 393(6684): 410.
[17] WATVEM S, SHEJVAL V, SONAWANE C, et al. The ‘K’ selected oligophilic bacteria: a key to uncultured diversity?[J]. Curr Sci, 2000, 78(12): 1535.
[18] 范一灵, 蒋波, 房蕊, 等. 药品无菌检查中微生物污染的鉴定和污染溯源分析[J]. 药物分析杂志, 2011, 31(6): 1067.
[19] 范一灵, 冯震, 钟玮, 等. 无菌药品生产企业核心区微生物污染调查与分析[J]. 中国药事, 2014, 31(6): 586.
[20] MARINELLI L, COTTARELLI A, SOLIMINI AG, et al. Evaluation of timing of re-appearance of VBNC Legionella for risk assessment in hospital water distribution systems[J]. Ann Ig, 2017, 29(5): 431.
[21] ZHANG J, YANG H, LI J, et al. Current perspectives on viable but non-culturable foodborne pathogenic bacteria: a review[J]. Foods, 2023, 12(6): 1179.
[22] RAY J. Microbiological monitoring of dialysis water systems-which culture method?[J]. J Ren Care, 2007, 33(2): 66.
[23] TADA Y, IHMORI M, YAMAGUCHI J. Oligotrophic bacteria isolated from clinical materials[J]. J Clin Microbiol, 1995, 33(2): 493.
[24] LAGIER JC, KHELAIFI AS, ALOUMT I, et al. Culture of previously uncultured members of the human gut microbiota by culturomics[J]. Nat Microbiol, 2016, 1: 16203.
[25] AHN Y, LEE UJ, LEE YJ, et al. Oligotrophic media compared with a tryptic soy agar or broth for the recovery of Burkholderia cepacia complex from different storage temperatures and culture conditions[J]. J Microbiol Biotechnol, 2019, 29(10): 1495.
[26] 李珏, 洪利娅, 王知坚, 等. 制药用水微生物限度检查新方法的研究[J]. 药物分析杂志, 2014, 34(2): 376.
[27] REASONER DJ, GELDREICH EE. A new medium for the enumeration and subculture of bacteria from potable water[J]. Appl Environ Microbiol, 1985, 49(1): 1.
[28] 宋明辉, 范一灵, 秦峰, 等. 激光解吸快速菌种鉴定与不同前处理方法对常见食源性致病菌菌种的鉴定分析[J]. 上海预防医学, 2020, 32(5): 375.
[29] 范一灵, 房蕊, 蒋波, 等. 微生物鉴定分型技术应用于医药企业微生物污染调查[J]. 中国医药工业杂志, 2010, 41(11): 810.
[30] TIDSWELEC C, BOONE K. Environmental and personnel monitoring programs-a risk-based case study of Cutibacterium acnes[J]. PDA J Pharm Sci Technol, 2020, 74(4): 408.
[31] 李涛. 空气微生物采样及发展趋势[J]. 中国卫生检验杂志, 2003, 13(5): 538.

 >
>