鲂属鱼类线粒体基因组的比较及其系统发育分析
时间:
摘要: 基于 GenBank 中团头鲂线粒体基因组全序列和三角鲂、厚颌鲂、广东鲂的部分线粒体基因组序列,设计引物扩增出三角鲂、厚颌鲂和广东鲂 3 种鱼线粒体基因组全序列,同时对 4 种鲂属鱼类线粒体基因组全序列进行了比较分析。结果表明,4 种鲂属鱼类线粒体基因组基因排列顺序完全相同,排列紧密,均包含 13 个蛋白质编码基因、22 个 tRNA、2 个 rRNA、1 个非编码控制区( D-loop 区) 和 1 个轻链复制起始区( OL 区) 。除 ND6 和 8 个 tRNA 在 L 链上编码外,其余的基因均在 H 链上编码。4 种鲂属线粒体基因组 13 个蛋白质编码基因中,均呈现出较强的 A + T 偏向性和 C 碱基偏好。全序列比对结果显示,共有 758 个变异位点,其中非简约性信息位点有 691 个,占总变异位点的 91. 16% ,简约性信息位点有 67 个,仅占总变异位点的 8. 84% 。22 个 tRNA 基因中只有 11 个存在种间变异,共 23 个变异位点,主要发生在 tRNA 三叶草结构的 TΨC 和 DHU 臂环上。13 个蛋白质编码基因中共检测出 626 个变异位点,这些变异主要发生在密码子第三位,占总变异位点的 82. 59% ,其中变异位点数最多的是Cyt b基因,达 84 个,其次是 ND 4 基因( 83 个) 。因此,Cyt b 和 ND4 基因可作为备选的分子标记,用于鲂属群体间的遗传学研究。基于 4 种鲂属鱼类线粒体基因组全序列构建的 ML 树和 BI 树均显示,三角鲂与厚颌鲂的亲缘关系最近,团头鲂与它们的亲缘关系相对较近,而广东鲂与前述 3 种鲂属鱼类的亲缘关系均较远。
关键词: 鲂属; 线粒体基因组; 变异位点; 分子标记; 系统发育
在脊椎动物中,线粒体基因组为双链闭合环状分子,长度为 15 ~ 18 kb。与其他脊椎动物一样,鱼类线粒体基因组核酸序列和组成比较保守,且排列紧密。根据碱性氯化铯密度梯度离心中 G + T 含量的高低将双链分为重链( H 链) 和轻链 ( L 链) [1],它们绝大多数由 13 个蛋白质编码基因 ( ATP6、ATP8、COX1 ~ 3、Cyt b、ND1 ~ 6 和 ND4L) 、2 个核糖体 RNA 基因( rRNAs: 12S rRNA 和 16S rRNA) 、22 个转运 RNA 基因( tRNAs) 、1 个非编码区( D-loop 区) 和轻链复制起始区组成。其中,除 ND6 基因和 8 个 tRNA 基因( tRNAGln、 tRNAAla、tRNAAsn、tRNACys、tRNATyr、tRNASer、 tRNAGlu、tRNAPro ) 在 L 链上编码外,其余的基因均在 H 链上编码[2 - 3]。线粒体基因组具有信息量丰富、分子量小、基因结构及排列简单、多个拷贝、物种内几乎不发生重排、无内含子、母系遗传、进化速率快等优点,是一个相对独立的复制单位,是生物学家研究系统发育进化的有力工具,也是惟一可以提供在基因组水平上进行研究的分子标记。在近十年间,线粒体基因组已经被广泛地应用于脊椎动物群体的系统发生学、比较和进化基因组学、群体遗传学、分子进化、保护生物学和系统发育树重建等方面的研究[4 - 5]。
鲂 属 ( Megalobrama ) 隶 属 鲤 形 目 ( Cypriniformes ) 、鲤 科 ( Cyprinidae ) 、鲌 亚 科 ( Culterinae) ,主要分布于我国以及越南和俄罗斯的一些地区,是我国重要的养殖鱼类之一。目前分类 研 究 认 为 鲂 属 鱼 类 包 括 4 种: 团 头 鲂 ( Megalobrama amblycephala ) 、三 角 鲂 ( M. terminalis) 、厚颌鲂( M. pellegrini) 和广东鲂( M. hoffmanni) [6 - 7]。
目前,关于鲂属鱼类线粒体基因组的研究仍停留在 D-loop、Cyt b、ND2、COX1 和 COX2 等不同区域水平,其主要原因是得到 mtDNA 全序列比较耗时耗力,而且还受到各种技术的限制[8 - 9]。然而,现在研究普遍认为较长的线粒体基因组序列能够得到较为准确的进化关系[10 - 11],而鲂属鱼类中只有团头鲂的线粒体基因组全序列已经被克隆出来。针对鲂属鱼类线粒体基因组全序列数据信息还不够完善的现状,本研究克隆了三角鲂、厚颌鲂和广东鲂 3 种鱼的线粒体基因组全序列,并结合从 GenBank 数据库中获得的团头鲂线粒体基因组全序列,对 4 种鲂属鱼类线粒体基因组进行比较及其系统发育分析,为鲂属鱼类群体的生物多样性的保护、遗传学研究及分子标记遗传育种等提供遗传信息,也为揭示鱼类线粒体基因组进化规律提供参考。
1 材料与方法
1. 1 数据来源
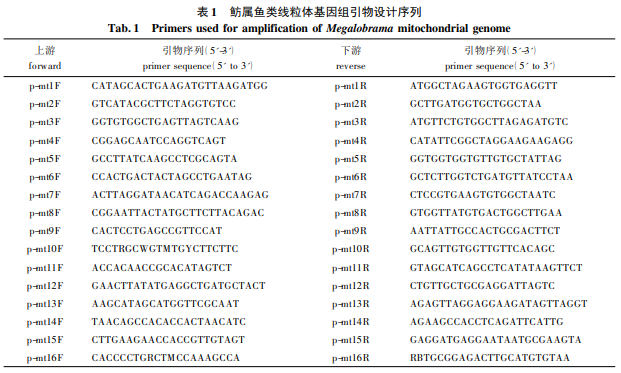
实验所用的三角鲂和广东鲂采集于珠江水系,厚颌鲂采集于长江上游的龙溪河。采集后的鳍条组织样品用无水乙醇固定,置于 - 20 ℃ 的冰箱中存储备用。总基因组 DNA 参照 Li 等[12]的方法用醋酸铵沉淀法从组织样品中提取。基于 GenBank 中团头鲂线粒体基因组全序列和三角鲂、厚颌鲂、广东鲂的部分线粒体基因组序列,用 Primer premier 5. 0 软件设计扩增了三角鲂、厚颌鲂和广东鲂 3 种鱼线粒体基因组全序列的 16 对引物( 表 1) ,其中,p-mt16F 和 p-mt16R 是参照黄志坚等[13]的方法所设计的简并引物。PCR 扩增产物用琼脂糖凝胶电泳检测,纯化后 T-A 克隆到大肠杆菌中,然后将菌液直接送往上海英骏公司进行测序。测得的序列在 NCBI 中用 BLAST 进行同源序列搜索,然后用 ClustalX 1. 83软件[14]和 BioEdit 7. 0软件[15]对测出序列进行比对、校正,人工拼接得到完整的线粒体基因组全序列。3 种鲂属 鱼 类 线 粒 体 基 因 组 全 序 列 已 经 提 交 GenBank,序列登录号分别为 JX242528、JX242529 和 JX242530。团头鲂线粒体基因组全序列从 GenBank 数据库中下载( 登录号 NC_010341. 1) 。
1. 2 碱基组成特征及密码子使用情况
用 MEGA 5. 10 软件[16]对 4 种鲂属鱼类线粒体基因组序列碱基组成特征及其密码子使用情况进行分析,并用 Perna 等[17]的方法进行 AT 和 GC 偏移度计算[AT-skew = ( A - T) /( A + T) 和 GCskew = ( G - C) /( G + C) ]。相对同义密码子的使用频率( relative synonymous codon usage,RSCU) 是衡量密码子使用偏好性的重要指标,它能直观地反映密码子使用的偏好程度[18]。它是用观察到的某一同义密码子的使用次数除以预期该密码子的出现次数,若 RSCU 的值小于 1 说明该密码子出现的次数比预期低,若 RSCU 的值大于 1 说明该密码子出现的次数比预期多[19]。
1. 3 变异位点分析
通过 ClustalX 1. 83 软件[14]对各基因进行多重序列比对。然后,通过 DnaSP 5. 0 软件[20]对 4 种鲂属鱼类线粒体基因组的主编码基因进行基因特征及变异位点分析。
1. 4 tRNA 基因二级结构预测
用 在 线 tRNAscan-SE 1. 21 软 件 和RNAstructure 5. 3 软件对 4 种鲂属鱼类发生变异的 tRNA 进行二级结构的预测,从而对它们的变异位点进行详细的定位分析。
1. 5 系统发育树构建
为探讨 4 种鲂属鱼类的亲缘关系,以翘嘴鲌 ( Culter alburnus) ( 登录号 NC _013616. 1) 和蒙古鲌( Culter mongolicus) ( 登录号 NC _008683. 1) 为外群,基于线粒体基因组全序列,使用 PhyML 3. 0 软件[21]的最大似然法( maximum-likelihood,ML) 和 M rBayes 3. 12 软 件[22] 的贝叶斯推论法 ( Bayesian inference,BI) 对 4 种鲂属鱼类进行系统发育树的构建。
2 结果
2. 1 鲂属鱼类线粒体基因组的基本特征
4 种鲂属鱼类线粒体基因组全序列长度为 16 621 ~ 16 623 bp ( 团 头 鲂 16 623 bp、三 角 鲂 16 621 bp、厚颌鲂 16 621 bp、广东鲂 16 622 bp) ,均编码 37 个基因( 13 个蛋白质编码基因、22 个转运 RNA 基因和 2 个核糖体 RNA 基因) 以及 1 个非编码 区 ( D-loop 区) 和轻链复制起始区 ( OL 区) 。除 ND6 和 8 个 tRNA ( tRNAGln、tRNAAla、 tRNAAsn、tRNACys、tRNATyr、tRNASer、tRNAGlu、 tRNAPro ) 在 L 链上编码外,其余的基因均在 H 链上编码。它们的基因排列顺序、长度、编码链、起始和终止密码子使用等方面非常相似( 表 2) 。13 个蛋白质编码基因和 22 个 tRNA 基因的排列顺序和长度完全相同。整个基因组排列紧密,仅仅只有少许的碱基间隔区( 13 处 33 bp,间隔碱基数为 1 ~ 13 bp) 和重叠区( 6 处 22 bp,重叠碱基数为 1 ~ 7 bp) 。在蛋白质编码基因序列中,有 4 对基因间存在开放阅读框的重叠,其中 ATP8 /ATP6 基因和 ND4L /ND4 基因间都存在 7 bp 的重叠,ND5 和 ND6 基因间存在 4 bp 的重叠,ATP6 /COX3 基因间存在 1 bp 的重叠。同时,相邻 tRNA 基因之间也存在有个别碱基的重叠现象,均有 2 处 3 bp 的重叠( tRNAIle /tRNAGln 2bp、tRNAThr /tRNAPro 1 bp) 。另外,鲂属鱼类线粒体基因组的 13 处间隔区中,最大间隔区位于 tRNAAsp和 COX2 之间,为 13 bp。基因间既没有重复又没有间隔的紧密排列基因对共计 21 处。
在蛋白质编码基因的起始和终止密码子方面,广东鲂的 ATP8 基因的终止密码子是 TAG,而其他 3 种鲂属鱼类均以 TAA 作为终止密码子,除此之外的 12 个蛋白质编码基因的起始密码子 ( ATG 或 GTG) 和 终 止 密 码 子 ( TAA、TA - 或 T - ) 在 4 种鲂属鱼类中完全相同( 表 2) 。
2. 2 鲂属鱼类线粒体基因组的碱基组成与偏好
对 4 种鲂属鱼类线粒体基因组及各组成部分碱基的平均组成及偏好情况进行了统计,结果显示,4 种鲂属鱼类线粒体基因组全序列 A + T 平均含量为 56. 0%,表明鲂属线粒体基因组全序列存在一定 A + T 偏向性( 表 3) 。鲂属线粒体基因组全序列还呈现出较强的 A 碱基和 C 碱基偏好( ATskew = 0. 12 和 GC-skew = - 0. 27) ,而且,4 种鲂属鱼类线粒体基因组的碱基组成还具有一定的规律性,除 OL 区序列和蛋白质编码基因第三密码子外,鲂属鱼类线粒体基因组的全序列、蛋白质编码基因、tRNA、rRNA 和 D-loop 区序列都存在一定的 A + T 偏向性、A 碱基偏好和 C 碱基偏好,其中 Dloop 区的 A + T 偏向性最为显著为 64. 2%,且明显高于线粒体基因组其他各组成部分的基因,因此 D-loop 区又称为 A + T 丰富区。
2. 3 鲂属鱼类蛋白质编码基因密码子使用情况
对 4 种鲂属鱼类的 13 个蛋白质编码基因的密码子平均使用频率和相对同义密码子平均使用频率进行了统计,结果见表 4。表 4 中粗体字显示的是编码同种氨基酸使用频率最高的密码子,这些密码子的 RSCU 值均大于 1,均为偏好密码子。结果显示,所有蛋白质编码基因的密码子使用都存在 着 强 烈 的 偏 好 性,其 中 NNA 密 码 子RSCU 基本上都大于 1,表明第三位点为 A 的密码子使用频率较高,密码子使用的这种偏好性与蛋白质编码基因的密码子第三位点的 A 偏好性一致。
2. 4 鲂属鱼类线粒体基因组序列比对
对 4 种鲂属鱼类线粒体基因组全序列、13 个蛋白质编码基因序列、12S rRNA、16S rRNA 和 Dloop 区的序列分别进行种间两两 BLAST 比对。
线粒体基因组全序列比对结果显示,团头鲂、厚颌 鲂 和 三 角 鲂 的 相 似 度 极 高 ( 98. 91% ~ 99. 66% ) 。其中,厚颌鲂和三角鲂的线粒体全序列相似度高达 99. 66%,团头鲂与三角鲂的相似度为 99. 06%,团头鲂与厚颌鲂的相似度也达 98. 91%。广东鲂线粒体全序列与其他鲂属鱼类的相似度为 95. 94% ~ 96. 05%。这表明三角鲂与厚颌鲂的亲缘关系最近,团头鲂与它们的亲缘关系相对较近,而广东鲂与前述 3 种鲂属鱼类的亲缘关系均较远。
线粒体不同结构区的序列比对结果与全序列比对结果相同,团头鲂、厚颌鲂和三角鲂 3 种间各结构区的序列相似度极高。厚颌鲂与三角鲂的序列相似度高达 99. 33% ~ 100% ,它们的 COX1 和 ATP8 序列完全相同,团头鲂与厚颌鲂和三角鲂的 ATP8 序列完全相同。在比对的 16 个线粒体结构区中,4 种鲂属鱼类 ND2 序列相似度最低,低于种间线粒体基因组全序列的相似度。另外,团头鲂、厚颌鲂和三角鲂 D-loop 区序列的种间相似度 ( 98. 30% ~ 99. 34% ) 略低于线粒体基因组全序列的相似度( 98. 91% ~ 99. 66% ) ,但这 3 种鲂与广东鲂 D-loop 区序列的相似度却略高于线粒体基因组全序列的相似度。
2. 5 鲂属鱼类线粒体基因组变异位点分析
4 种鲂属鱼类线粒体基因组全序列比对结果显示,共有 758 个变异位点,其中非简约性信息位点有 691 个,占总变异位点的 91. 16% ,简约性信息位点有 67 个,仅占总变异位点的 8. 84% 。其中由广东鲂单独变异引起的单现变异位点数就有 571 个,占 4 种鲂属鱼类的单现变异位点数的 82. 6% ,可见广东鲂与其他 3 种鲂属鱼类( 团头鲂、三角鲂和厚颌鲂) 的亲缘关系比较远。简约性信息位点数最多的是 ND2,其次为 ND1、COX1 和 Cyt b,鲂属鱼类线粒体基因的种间分化主要体现在这 4 个基因中( 表 5) 。ND4L,ATP8 和 COX2 及 18 个 tRNA 序 列 ( 除 tRNAPhe、tRNAGlu 和 tRNAGly外) 中均无简约性信息位点。
4 种鲂属鱼类线粒体基因组 13 个蛋白质编码基因和 2 个核糖体 RNA 基因的变异位点的分析结果显示,12S rRNA 和 16S rRNA 两个核糖体RNA 基因的保守性最高,变异位点比例分别仅为 2. 29% 和2. 25% ; 其次是 ATP8 和 COX1 基因( 分别为3. 03% 和 3. 35% ) 。ND2 基因变异位点的比例最高达到 7. 46% ; 然后是 Cyt b 基因( 7. 36% ) 。变异位点数最多的基因为 Cyt b 基因,达 84 个; 然后是 ND4 基因( 83 个) ( 表 5 粗体字表示) 。
2. 6 不同基因中变异位点的分布
蛋白编码基因中变异位点的分布 4 种鲂属鱼类线粒体基因组 13 个蛋白质编码基因密码子不同位点的变异分析显示,变异位点主要发生在密码子第三位点,占总变异位点的比例为 60. 00% ~ 92. 86% ; 其次发生在密码子第一位点,占总变异位点的比例为 3. 85% ~ 40. 00% ; 而密码子第二位点最为保守,只 有 COX1、ND1、ND2、 ND5 和 ND6 基因发生了变异,仅占总变异位点比例的 0 ~ 7. 69% ( 表 6) 。4 种鲂属鱼类 COX1 ~ 3 的 3 个亚基在密码子第三位点发生变异比例都很高,最 高 的 是 COX2 ( 92. 86% ) ,其 次 是 COX1 ( 92. 31% ) ,最低的是 COX3( 91. 89% ) 。
4 种鲂属鱼类线粒体基因组的蛋白质编码基因中,由于碱基的转换或颠换,引起氨基酸序列变化的位点数共有 52 个,且 13 个蛋白质编码基因都存在着氨基酸的变异,其中 ND2 基因中的氨基酸变异比例最高,达到 4. 02% ,其次是 ATP8 和 ND3 基因,而 COX1、COX2、COX3 和 ND4 4 个基因的氨基酸变异比例都很低( 表 7) 。
转运 RNA( tRNA) 中变异位点的分布 4 种鲂属鱼类均编码 22 个 tRNA 基因,其中除了 4 个 tRNA[tRNALeu ( UUN) 、tRNALeu ( CUN) ; tRNASer ( UCN) 、tRNASer( AGN) ]含有 2 个外,其余 18 个 tRNA 均只有1 个,它们的排列顺序和长度完全一致,长度从 68 ~ 76 bp 不等( 表 2) ,说明 4 个物种不存在基因重排,序列相似度高。用在线软件 tRNAscan-SE 1. 2. 1 和 RNA structure 5. 2 软件预测 4 种鲂属鱼类均编码的 22 个 tRNA 基因的二级结构,其二级结构也基本一致。除了 tRNASer ( AGN) 缺少二氢尿嘧啶臂( DHU stem) ,在相应的位置上只形成一个简单环外,其余的 tRNA 基因都形成典型的三叶草结构。22 个 tRNA 中只有 11 个 tRNA 相对比较保守,未发生变异( 表 5) ,剩余 的 11 个 tRNA ( tRNACys、tRNAGlu、tRNAGly、 tRNA His、tRNAIle、tRNA M e、tRNAPhe、tRNAPro、 tRNAser( AGN) 、tRNATyr 和 tRNAVal ) 均发生了变异 ( 表 8) 。从表 8 可以看出,这 11 个 tRNA 总共有 23 个变异位点,主要分布在三叶草结构的 TΨC 和 DHU 臂环上,各分布着 8 个变异位点,反密码子臂环上只分布有 4 个变异位点,剩余 3 个变异位点分别分布在氨基酸接受臂上( 2 个) 和可变环上( 1 个) 。
OL 区和 D-loop 区中变异位点的分布 4 种鲂属鱼类的轻链复制起始区( OL 区) 位于 WANCY 区域( tRNATrp-tRNAAla-tRNAAsn-tRNACys-tRNATyr ) 的 tRNAAsn与 tRNACys之间,长度为32 bp,这段序列有一段回文序列,可折叠成茎 - 环结构。OL 区的 1 个变异位点发生 loop 环上( G?A) 和茎区相对保守的特征与脊椎动物 OL 区序列 loop 环变异较大、茎的长度和组成均十分保守的特征相一致[23]。
4 种鲂属鱼类线粒体基因组中,非编码区( Dloop 区) 位于 tRNAPro和 tRNAPhe基因之间,与其他硬骨鱼类的一致。4 种鲂属鱼类 D-Loop 区通过与其他鲤科鱼类相比较,均识别出 1 个终止序列区 ( TAS) 、3 个中央保守区( CSB-F、CSB-E 和 CSB-D) 和3 个保守序列区( CSB1、CSB2 和 CSB3) 。TAS 有 4 个变异位点位于其中,是 D-loop 区中序列变异最大的区域; CSB-F 和 CSB-D 较 CSB-E 序列更为保守,没有变异,CSB-E 存在一个碱基 A 的插入; CSB1 和 CSB2 序列完全相同,显示出该序列的保守性,而 CSB3 序列存在一个碱基的转换( A?G) 。
2. 7 系统发育树分析
以翘嘴鲌和蒙古鲌 2 种鲌属鱼类作为外群,分别采用最大似然法和贝叶斯法构建了 ML 树和BI 树。系统发育树各分支的置信度以自举检验值( ML 树) 和后验概率( BI 树) 来表示。ML 树和 BI 树的拓扑结构是完全相同的,而且它们的置信度都极高( 图 1) 。系统发育树的结果显示: 三角鲂和厚颌鲂首先聚在一起,然后与团头鲂聚在一起,最后与广东鲂聚在一起,形成一支( 鲂属) ,表明三角鲂与厚颌鲂的亲缘关系最近,团头鲂与它们的亲缘关系相对较近,而广东鲂与前述 3 种鲂属鱼类的亲缘关系均较远。——论文作者:赖瑞芳1 , 张秀杰1 , 李艳和1 , 吴俊颉1 , 杨东辉2 , 王卫民1*
本文来源于:《水产学报》(月刊)创刊于1964年,是由中国水产学会主办、上海海洋大学承办的以水产科学技术为主的学术性刊物。主要刊载水产基础研究、水产养殖和增殖、渔业水域环境保护、水产品保鲜加工与综合利用、渔业机械仪器等方面的论文、研究简报和综述。

 >
>