单细胞技术与干细胞疗法在心脏疾病研究中的应用
时间:
摘要细胞是生命的基本单位,而异质性则是细胞的固有属性之一。单细胞测序技术的出现,为细胞间异质性的探索、新的细胞群的发现、发育轨迹重建的研究提供了便利。目前,单细胞测序技术已在包括心肌梗死、心肌肥大、心力衰竭在内的研究中有了一定的成果。该文综述了单细胞测序的流程,包括样品制备、文库构建和测序分析,回顾了其在心脏疾病中的应用,并讨论了基于干细胞的疗法在临床中的应用前景。
关键词单细胞测序技术;干细胞疗法;心脏疾病
心脏疾病一直是世界范围内致死率最高的疾病之一[1]。其主要原因是哺乳动物在发育过程中,心脏逐渐丧失再生修复的能力。各种病理损伤导致心脏细胞不可逆的损失,也就使心脏疾病的治疗面临极大的挑战。干细胞技术的突飞猛进使得在体外获得各类心脏细胞成为可能,干细胞移植、心脏“补丁”和心脏类器官等一系列基于干细胞的研究也为心脏病的临床治疗提供了新思路。细胞间的变异和异质性是干细胞的基本特征,在进行多组学分析时,如果用bulk的手段,这些差异将被掩盖[2]。而单细胞RNA测序(single-cellRNAsequencing,scRNA-seq)技术可以深入剖析细胞异质性,识别不同类型的细胞,在“同质”的干细胞群体中也不例外[2]。近年来,单细胞质谱流式技术(cytometrybytimeofflight,CyTOF)和scRNA-seq技术被越来越多地用于心血管领域,可以预见这将是未来研究心脏疾病的一大趋势。本文主要总结了以scRNA-seq为代表的单细胞组学技术在心脏相关病症以及干细胞相关疗法中的应用,期望能为心脏病的发病机制、疾病模型构建、药物筛选等提供参考。
1单细胞测序技术
自20世纪90年代DNA微阵列(DNAmicroarray)出现以来[87],转录组学技术的革新推动了各个领域研究的进程。2009年,TANG等[2]建立了最初的scRNA-seq流程,随后多种单细胞转录组学平台技术涌现,如标签测序法STRT-seq[24]、CEL-seq[21]、CELseq[22];全长测序法SMART-seq[24]和SMART-seq2[25-26]等。这些方案在扩增方式、转录本覆盖度以及相应平台的选择方面略有不同,但都已广泛应用于包括心脏病在内的各种疾病的研究中。
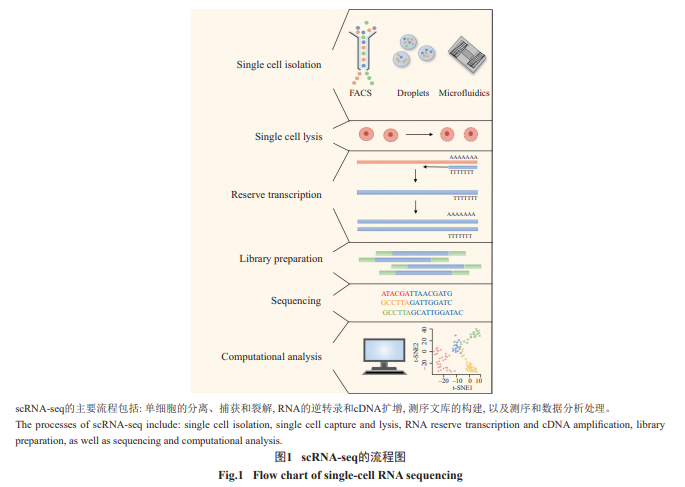
scRNA-seq是将分离的单个细胞的全基因组DNA扩增,获得完整基因组后进行高通量测序的一种组学技术,通常用于研究细胞的异质性。目前,scRNA-seq主要包括如下几个步骤:单细胞的分离、捕获和裂解,RNA的逆转录和cDNA扩增,测序文库的构建,以及测序和数据分析处理(图1)。根据实验设计和目标的不同,进行scRNA-seq各个步骤所用的技术和方法也不尽相同[3-6]。
1.1单细胞样品的制备和单细胞的捕获
单细胞样品的制备是进行所有单细胞研究的第一步。而使用酶和机械分离的方法从致密的组织材料中分离细胞,是后续组学分析中引起变异或错误的主要因素。所以,研究人员必须确定进行组织处理、消化和细胞分选的相应方法,以确立最优化的制备流程[7]。多能干细胞用经典的消化方式:胰蛋白酶、EDTA或商品化的消化液(如accutase)即可完成单细胞样品的制备,而从悬浮液(如血液)中分离得到的细胞,只需简单将其洗净,即可进入后续的scRNA-seq流程。在诸如脾脏或淋巴结之类的一些软组织中,一般无须进行酶消化,可以直接通过机械手段(如70μm尼龙网)获得单细胞悬浮液,继而从中分离出淋巴细胞[7]。但是对于其他组织来源的细胞,通常需要一定的处理才能获得单细胞悬液。在人体心脏组织的单细胞分离中,常采用在消化处理前先对组织块进行切片的方法[8],以获得高质量的成人心肌细胞进行单细胞转录组学分析[9]。
现今,手工分选捕获单细胞已经不是主流方式,根据不同的实验目的,捕获手段是多种多样的,如激光捕获、荧光激活细胞分选(fluorescence-activatedcellsorting,FACS)、微液滴和微流控等[10-11]。对于非心肌细胞和未成熟心肌细胞的单个心脏细胞捕获,其流程与其他体细胞相似,但成年人心肌细胞形状细长且细胞体积较大(平均直径>100μm),某些病理状态下甚至更长更大,限制了它们与许多现有单细胞平台比如微流控和液滴系统的相容性[13-14]。最初,心肌细胞的转录组水平研究是通过手动移液将单个细胞移入多孔板中[14-15],这些操作不仅费时费力而且也存在着一定的偏差。而基于微流控芯片技术ICELL8单细胞分选平台的出现为心肌细胞(CMs)的捕获提供了新的可能性,其芯片具有5184个纳米孔,一次最多可以分离1800个单细胞[16],具有高通量低成本的优势。目前认为,基于FACS的捕获方法是比较好的选择之一。传统的FACS因其较快的流体分选过程,可能会使细胞产生一定的损伤,造成了捕获和后续分析上的困难[15]。研究人员发现,使用大颗粒荧光激活细胞分选(largeparticleFACS,lp-FACS),通道尺寸为500μm),可以成功分离出细胞形态良好、RNA质量较高的成年小鼠心肌细胞[15]。然而,在一些情况下很难实现对细胞的“无伤”分离。对于一些临床样本,比如经历过像移植这种长时间的流程后取下的组织样本,或是从尸体上采集的样本,单核转录组测序(single-nucleusRNAsequencing,snRNAseq)技术则是一个更好的选择。该方法利用了细胞核在反复冻融过程中的抗降解能力,同时也可以规避因细胞体积过大而产生的捕获困难[17-19]。利用此项技术SEE等[18]发现,长基因间非编码RNA(longintergenicnon-codingRNA,lincRNA)是调控小鼠CMs细胞周期的关键因子。虽然snRNA-seq作为scRNAseq的补充具有一定的优势,但是其局限性也不可忽视。已知大多数基因主要定位于细胞质中,但仍有一部分基因在细胞核中有更多的转录本[19]。使用snRNA-seq进行分析时,这些核富集的转录本可能会导致细胞周期、转录和泛素周期等生物过程被过度强调[20]。因此,仅测量核mRNA的表达也可能会导致基因转录本相对丰度的失真,影响细胞类型识别和细胞聚类。相信随着单细胞水平研究的不断深入,根据不同的实验设计和目的需求,相应的平台技术流程也会随之优化和进步。
1.2单细胞基因组的扩增与文库的构建
在成功分离和捕获目的细胞之后,需要生成相应的文库,即在裂解细胞获得RNA后将其反转录为cDNA。值得注意的是,由于细胞中mRNA的含量一般较低,需要在逆转录后对cDNA进行预扩增,通常通过PCR扩增[24],或是多重线性扩增(multiplexedlinearamplification)如CEL-seq[21]、CEL-seq2[22]、STRTseq[23]、SMART-seq[24]、SMRT-seq2[25-26]、DROP-seq等来实现(表1)。SMART-seq2等线性扩增的方式能够有效地解决PCR指数扩增造成的偏差,同时具有较高的灵敏度和较好的文库产量,也在降低成本的基础上缩短了扩增时间[25-26]。在有大量细胞分析需求时,DROP-seq则是很好的选择,其通过微流控装置将单个细胞包裹在带有DNA条形码微珠的纳升级液滴中,这些条形码随后会连接到逆转录的cDNA上,由此对数以千计的细胞进行平行分析[86]。已有研究团队利用Drop-seq对小鼠胚胎干细胞(mouseembryonicstemcells,mESCs)进行细胞群解构分析,发现在白血病抑制因子(leukemiainhibitoryfactor,LIF)戒断后细胞分化的异质性[29]。近年来,出现了将Drop-seq和10×GenomicsChromium相结合的方法(Droplet-basedsequencing),其通过微流体墨盒形成的纳升级液滴,将单个细胞置于油基介质中,以分离细胞进行微反应,这是目前最高通量的方法,一次可评估成千上万个细胞[27-29]。但是,基于Drop-seq的扩增手段也存在着一定的局限,比如读取深度较浅,无法对可变剪切等进行深入的研究。目前认为,其更适合鉴定罕见的细胞群,或研究整个器官水平的细胞多样性。
在构建文库的过程中,一般存在着mRNA的降解和反转录效率不足的问题,仅有10%~20%的转录产物能够成功合成cDNA[28,31-32]。非均一的扩增就导致某个特定转录本的读取次数并不能代表样本中转录本的数量,ISLAM等[33]通过唯一分子标识符(uniquemolecularidentifiers,UMIs)解决了这一问题,附在单个cDNA分子上的UMIs可以有效地消除扩增偏倚。同样,在合成转录本时加入已知浓度的人工生成的mRNAs(RNAspike-ins),也是校准系统误差,达到标准化分析的选择之一[34]。
1.3单细胞测序分析
第二代测序(nextgenerationsequencing,NGS)技术的问世,以及适用于单细胞“包装”的微流控设备和多孔板的出现,共同推动了单细胞转录组学的发展[28,30]。NGS又称为高通量测序技术(high-throughputsequencing),是基于PCR和基因芯片的DNA测序技术,与传统的Sanger测序相比,NGS减少了对DNA模板的需求量,提高了读取的可靠性,降低了RNA测序的成本并缩短了测序时间。而样品良好的质量控制则是后续分析结果的保证,采取一些预处理质量控制步骤来过滤低质量的细胞是至关重要的,能够有效避免对数据的错误解读。线粒体reads则是目前较为公认的质量控制指标,一般线粒体含量超过30%的细胞被认为是应激或死亡细胞,应从后续分析中剔除。但是这一标准也应根据不同组织和细胞类型进行调整,比如心肌细胞的线粒体含量就显著高于其他组织细胞。研究发现,在人类心脏组织样本中,线粒体转录本约占总mRNA的30%[35],而在其他能量需求较少的器官中,这一比例约为5%或更低[36]。GLADKA等[37]报道,在成年小鼠心肌细胞中,心肌细胞的线粒体转录本比率(58%~86%)明显高于其他类型的细胞。因此,在处理心肌细胞的单细胞数据时,需要进一步研究线粒体DNA比例与心肌细胞状态之间的关系,以规范这一质量控制指标。
目前,scRNA-seq数据一般通过降维的手段去除噪声、简化样本之间的变化,使多基因(数十个或几百个)之间的数据差异可视化。诸如tSNE(t-distributedstochasticneighborembedding)、UMAP(uniformmanifoldapproximationandprojection)、PCA(PrincipalComponentAnalysis)等算法可以将复杂数据简化生成二维模型以达到可视化的目的[7]。对这些降维后的数据随后便会根据需求进行下游分析,包括差异基因表达、上下游信号通路富集、轨迹分析等[38]。Monocle是进行单细胞轨迹分析的有力保障,通过单细胞转录组的表达矩阵,利用无监督算法将细胞归位于不同的发育轨迹分支上,从而模拟细胞群体生物学过程。其能够将细胞按照模拟的时间顺序进行排列,展现包括细胞分化等生物学过程[39]。基于Monocle的Monocle2则是目前应用最为广泛的分析手段,可用于所有的scRNA-seq数据集,并且不需要有关细胞命运或有关分支点的信息输入,即可模拟出时间发育过程的动态变化[40]。一般来说,Monocle2首先通过t-SNE选取在细胞群间差异表达的基因,随后使用dpFeature算法进行密度峰聚类,以半监督的分析模式,针对目的细胞亚群进行个性化分析。2019年,CAO等[73]研发出Monocle3,其利用UMAP进行降维,针对小鼠器官发生过程,重建了56条不同细胞亚型的轨迹。现在,将scRNA-seq与各种算法相结合是一个常规的研究方法,该流程可以针对不同的研究目标,从单细胞水平鉴定稀有的细胞类型,评估细胞的异质性,同时也能阐明细胞的动力学特征并揭示细胞间的交互作用。
2单细胞测序技术在干细胞和心血管研究领域的应用
2.1单细胞技术对干细胞研究的意义
多能干细胞(pluripotentstemcells,PSCs)是包括胚胎干细胞(embryonicstemcells,ESCs)和诱导多能干细胞(inducedpluripotentstemcells,iPSCs)在内的一类具有多能性和自我更新能力的细胞。PSCs的这些特质也为了解早期胚胎发育和再生医学、药物筛选提供了无限的可能性。同时,PSCs的异质性也是其基本特性之一,即在一种细胞类型中,转录组也会随着信号激活、细胞周期、细胞凋亡、应激或昼夜节律的改变而变化[45]。传统的“块样”RNA测序(bulkRNA-seq)分析手段,因为每个基因的最终表达值是大量细胞表达水平的平均值,掩盖了细胞间基因表达的细微差异或遗漏了一些特异性的细胞亚群。而诸如单分子荧光mRNA原位杂交和单细胞qPCR在检测基因的表达差异方面,虽然其灵敏度相对较高也比较可靠,但也有着低通量的局限性,仅适用于分析较少的目标基因表达情况[44]。scRNA-seq则可以体现出这些差异,并准确地识别出各种细胞亚群[43]。早在2016年,已有团队通过scRNA-seq揭示了造血前干细胞的核心分子特征,并阐明了造血干细胞分化的逐步机制[46]。scRNA-seq能够以无偏倚的方式对干细胞的异质性、细胞间基因表达的细微差异进行剖析,其高通量、高灵敏度的优势,是干细胞领域分析的一大利器。
2.2干细胞在心血管疾病领域的应用
1998年,人类胚胎干细胞(humanembryonicstemcell,hESC)系由THOMSON[47]首次建立,随后各种诱导干细胞向心肌细胞定向分化的方案涌现,无论是在疾病模拟、病理学研究还是再生医学等方面都得到了广泛的应用(图2)。基于干细胞的细胞疗法是当下研究的热点。研究表明移植人胚胎干细胞来源的心肌细胞(hESC-CMs)可有效改善心肌梗死后大鼠和猪的心脏功能[48-50],CHONG等[51]也证实,在非人类灵长类动物的心梗模型中,外源性移植hESC-CMs可以使梗死区域重新肌肉化。最初的细胞疗法,一般是向心肌受损部位直接注射hESCCMs,以代替心肌损伤后死亡的细胞,但这一方法存在着诸多的问题,比如难以精确控制注入细胞数量、难以到达治疗的靶位点、注入细胞的存活率较低等[53-54]。
然而,基于hESCs的干细胞疗法一直存在着伦理学相关和免疫排斥的问题,也就局限了其在临床治疗领域的应用。直到2006年,YAMANAKA等[52]发现,小鼠成纤维细胞可以通过转录因子Oct4、Sox2、c-Myc和Klf4重新编程为iPSCs,为攻克这些难题提供了新的可能性。iPSCs被认为是自体的,不需要免疫抑制,且病人体细胞来源的iPSCs可以通过基因编辑纠正致病性突变或变异,进一步帮助逆转疾病表型,也就为基于干细胞的个体化治疗提供了可能。随着组织工程学的不断发展,使干细胞来源的心肌“补片”逐渐取代了注射疗法[53]。将具有高纯度(近90%)心肌细胞的“补片”移植至心肌梗死12周猪的模型中,在后续8周的观察中,发现其能够减轻左心室的重构,增加血管形成,并抑制纤维化[55]。近期也有研究表明,移植临床尺寸(4cm×2cm×1.25mm)的人心肌“补片”可显著改善心肌梗死猪的左心室功能,有效缩小梗死面积[56]。迄今,这些疗法在心功能方面带来的改善一般是短期的,从几周到几个月不等。因此,产生表征更为成熟的、血管化的“补片”,以达到稳定且持久恢复心功能的目的将是今后研究的方向。
通过构建动物模型模拟各类心脏疾病的发生是当下主流的研究方式之一,但其存在成本较高、通量较低等短板。传统的病理模型构建大多是基于小鼠、家猪或是猕猴等模式动物,物种间存在着的生理差异,为人类心脏疾病的研究带来了一定的困难[57]。而人源性疾病模型独具优势,更为贴近人类疾病生理条件下的真实情况,也就扫除了研究疾病发生机制道路上的阻碍,为新的临床疗法的出现铺平了道路[58]。心脏疾病的致病因素之一是基因突变[59],通过干细胞技术直接使用患者的体细胞来模拟遗传心脏病则为其治疗提供了有力的支持。基于PSC的模型的建立,不仅为心脏疾病的发病机制研究提供了便利,也被广泛地应用于心血管新型药物的研发、药理学和毒理学预测等领域。MILLS等[76]利用基于hPSC-CMs构建的心脏类器官,已经成功筛选出了可以促进人心肌细胞增殖,同时对心律和收缩力影响较小的化合物。
相关知识推荐:国内与细胞相关的核心期刊有哪些
心脏由多种细胞构成,包括心肌细胞和其他支持细胞,如内皮细胞、成纤维细胞和免疫细胞等,细胞间的相互作用和多方面的调控是维持心脏稳态的关键。因此,使用相对不成熟的iPSC-CMs来构建疾病模型仍存在着很多不足。近年来,3D生物打印和3D冲压技术的发展,共同推动了类器官、器官芯片[75]、EHTs(engineeredhearttissues)[74]的产生,而通过这些方式模拟疾病的发生或是探索发育过程已成为当下研究的热点。诸如Barth综合症相关的心肌病[60]、Duchenne肌肉营养不良症[61]、原发性高血压诱发的左心室肥大[62]等病症都已通过上述方法成功建模。近期,NADAV等[63]更是通过3D打印技术,成功用人体细胞制造出世界上首颗3D打印心脏。同年,具有更加完整的微血管系统、能够收缩的微心脏的面世[64],更是鼓舞着世界范围内研究人员在这一领域不断探索。
2.3单细胞测序技术在基于干细胞疗法的心血管领域的应用
细胞聚类是目前scRNA-seq最广泛的应用之一,其在揭示细胞间的异质性、定义罕见的细胞群以及进行轨迹重建等方面发挥着不可替代的作用。研究人员通过对妊娠中期(E9.5)到成年期(P21)的小鼠心脏细胞进行scRNA-seq分析,在单细胞水平上以时空分辨率进行聚类,描绘了包括心脏发育和成熟等过程在内的谱系特异性转录调控图谱[67]。2012年,QIAN的团队[85]首次开发出直接将心脏中的成纤维细胞(fibroblasts,FBs)转化为类似于内源性心肌细胞的诱导性心肌细胞(inducedcardiomyocytes,iCMs)的方法,给干细胞疗法带来了福音。但这种转化事件并非一蹴而就的,是一个“异步”过程,即在重编程过程的各个阶段,细胞群体是由未重编程的细胞、部分重编程细胞和完全重编程细胞构成的。而这一过程中的异质性,在scRNA-seq的帮助下得到了有效的探索。LIU等[68]利用微流体单细胞RNA测序技术,发现了从FBs到CMs细胞命运转化期间的关键分子和主要信号通路,并成功地重建了该过程中FBs的转化轨迹。已有研究证实,过渡细胞的状态可以用“拟时态”排序来识别,Nkx2-5+和Isl1+的心脏祖细胞(cardiacprogenitorcells,CPCs)在小鼠发育过程中的轨迹表明,虽然Nkx2-5+CPCs遵循一条连续的发展路径,但Isl1+CPCs的轨迹却分叉成两个不同的谱系,也就证明其处于过渡状态,这和基因敲除实验以及后续分析的结果相吻合[41]。同时,轨迹推断也可以用作验证依据,SOYSA等[42]利用“拟时态”轨迹验证了他们关于细胞群异质性的假说,并证实了转录因子Hand2在流出道中的作用。在发育学研究中,科研人员通过“拟时态”排序,分别在心室和心房CMs中鉴定出三组基因,辅以基因共表达网络分析发现了发育过程中调控人类心脏早期和晚期发育基因表达的核心因子[69]。
近期,单细胞测序和谱系追踪技术的发展使得单细胞层面的谱系追踪成为可能。传统的追踪手段包括利用酶、荧光蛋白、病毒条形码等在内的手段标记细胞和其子代[70],而随着CRISPR-Cas9技术的兴起以及Cre介导的基因重组的应用,谱系追踪领域又有了新的突破。ALEMANY等[71]利用CRISPRCas9技术,对DNA双链断裂以插入或删除的方式进行修复,形成了独特的、可遗传的细胞标记,成功确定了斑马鱼胚胎发育过程中的克隆起源和细胞类型。同样,基于慢病毒的细胞标记(Celltagging)也是研究细胞命运不可或缺的手段之一,比如利用慢病毒研发的LARRY(lineageandRNArecovery),通过给每个细胞打上独一无二的“条形码”,实现对细胞状态和命运的同步追踪,研究者已将其应用于造血系统中命运决定的研究,并鉴定出两条单核细胞分化路线[72]。干细胞是目前研究胚胎发育的主要材料,而将这些新型的谱系追踪手段引入到发育研究或是绘制心肌细胞重编程过程的详细图谱中去,必将为深入了解其中分子机制、辨明轨迹分叉点、理解分子网络的运作等带来希望。
正如上文所述,成年人的心肌细胞与微流控平台存在着一定的不兼容性,在健康的心脏中,单个心肌细胞的长度可以达到150μm,在某些疾病情况下甚至更长,也就局限了单细胞水平CMs的研究。但是对于小鼠胚胎干细胞来源的心肌细胞(mESCCMs)或是hESC-CMs却没有这一局限,也就促使多能干细胞来源的CMs正在成为高通量药物筛选过程中的核心力量。在内皮素-1(hiPSCmyh7-CMs)构建的的心脏肥大模型中,利用高通量的药物筛选方法发现,维拉帕米治疗可以有效地缓解心肌肥大造成的损伤[65]。SHARMA等[66]研发了一种生成hiPSC-CMs的方法,通过对这些细胞进行收缩能力和相应的细胞毒性试验完成了其安全性评估。前文中提及的EHTs,也可以用于测量心肌功能的所有基本参数,如收缩力、传导速度、搏动频率、舒张压、被动张力和细胞内Ca2+瞬变等[77]。我们认为,将EHTs与组织学分析、蛋白质检测和测序技术(如scRNA-seq)相结合,实现多组学平台共同研究势必成为未来的研究热点。——论文作者:凤琦1,2王利1,2*

 >
>