豨桐丸治疗类风湿性关节炎的分子作用机制研究
时间:
摘要:目的:基于分子对接与网络药理学探究豨桐丸治疗类风湿性关节炎(rheumatoidarthritis,RA)的潜在生物学机制。方法:运用中药系统药理学数据库与分析平台结合文献挖掘,收集豨桐丸所含有的活性成分,并进行口服利用率、血脑屏障透过率和药物相似性等参数筛选;选择与RA相关的靶蛋白建立受体分子库;应用分子对接进行药物成分的初步筛选并通过50ns的分子动力学验证对接结果的可靠性;通过化学成分靶点预测和靶点注释,采用Cytoscape3.6.1软件进行活性成分靶点网络和靶点-疾病网络的构建及拓扑分析,验证了重要的成分与靶点;运用DAVID数据库对豨桐丸的靶点进行GO分析和KEGG富集分析。结果:豨桐丸中化学成分豆甾醇、羽扇醇、谷甾醇、芹菜素-7-O-β-D-葡萄糖醛酸苷丁酯主要通过疏水作用与相关靶蛋白结合发挥药效;豨桐丸治疗RA的机制涉及细胞过程、炎症反应过程等生物过程,涉及细胞膜、突触前膜、膜筏等细胞组分,白蛋白、肿瘤坏死因子、血管内皮生长因子A、丝裂原激活的蛋白激酶、前列腺素G/H合成酶2等多种物质;涉及的信号通路有神经活性配体受体相互作用通路、癌症蛋白多糖通路、人巨细胞病毒感染通路、JAK信号通路、乙肝通路等。结论:豨桐丸治疗RA的分子机制可能是通过多成分、多靶点、多通路的协同作用来发挥疗效,其主要生物学机制可能与神经活性配体-受体相互作用等信号通路有关,这为豨桐丸的推广及RA新药的研发奠定了研究基础。
关键词:豨桐丸;类风湿性关节炎;活性物质;分子对接;分子动力学;网络药理学
类风湿性关节炎(rheumatoidarthritis,RA)在中医学中称为“痹证”,是由于人体自身正气不足受到外邪后,气、血、津液、经络凝滞不通而导致的[1-10]。尽管现有药物对RA的疗效确切,但不良反应明显[11]。因此,探寻有效且不良反应小的中药方剂具有重要意义。《济世养生集》卷三中记载豨桐丸具有“祛风胜湿、舒筋活络之功效。主治感受风湿、两足酸软、步履艰难、状似风瘫”。该方由臭梧桐叶及豨莶草等量调剂而组成。《新修本草》中记载“豨莶草,辛、苦、寒,有祛风湿、利关节、解毒之功效”;《本草图经》中记载“臭梧桐叶,药性辛、苦、甘、凉,归肝经,祛风湿、通经络、平肝”。现代临床应用中,豨莶草常被应用于治疗风湿痹痛、中风半身不遂、四肢麻木、腰膝酸软[12]等;臭梧桐叶被应用于治疗风湿痹症[13],两者相须为用,临床主要用于治疗风湿、RA及其相关病症,尚未有不良反应的报道。研究表明,豨桐丸通过抑制炎性介质来治疗RA[14],但无分子水平上的治疗机制研究。本研究运用分子对接、分子动力学,结合网络药理学研究豨桐丸活性成分与RA相关靶蛋白的结合模式和结合能,探究豨桐丸治疗RA的物质基础,通过单成分-多靶点、多成分-单靶点、单成分-单靶点的作用分析,探讨了豨桐丸活性成分与RA相关靶蛋白的作用机制,从豨桐丸的活性成分中筛选出可能用于治疗RA的关键成分,为进一步的药物设计与研发提供理论基础。
1材料与方法
1.1豨桐丸活性成分数据库的建立臭梧桐叶的提取物含有多种化学成分。王昭[15]把臭梧桐叶的化学成分分为六类:挥发油类,共5种,分别为1-己醇、伽罗木醇、2,6,10,15-四甲基十七烷、5-甲基-3-庚酮和2,6,6-三甲基环己酮;黄酮类,共8种,分别为芹菜素、芹菜素-7-葡萄糖醛酸、车前草苷、臭梧桐素、海常素、海州常山苷、刺槐素-7-二葡萄糖醛和金合欢素-7-二葡萄糖醛酸;还有苯丙素类、生物碱类、糖苷类和其他类。宋婷[13]、KimKM等[16]发现,豨桐丸中含有的苯丙素类化合物有异青角甾苷、毛蕊花糖苷和去咖啡酰毛蕊花糖苷,还发现了其他苯丙素类化合物咖啡酸甲酯、去鼠李糖洋丁香酚苷、去鼠李糖异洋丁香酚苷、3,4-二羟基苯乙醇[17]、1-O-咖啡苷、洋丁香酚苷、异洋丁香酚苷、地黄苷和异地黄苷[18];生物碱类化合物有常山碱、常山定碱和异常山定碱[19]。胡海军等[20]采用石油醚对臭梧桐叶进行萃取得到了羽扇豆醇、木栓酮、白桦脂酸、蒲公英赛酮、赪桐甾酮等有机物质。徐瑞兰等[21]从臭梧桐叶提取分离了11种化学成分:β-谷甾醇、β-胡萝卜苷、芹菜素、芹菜素-7-O-β-D-葡萄糖醛酸苷丁酯、乙酸橙酰胺、类叶升麻苷、马替诺皂苷、5-羟甲基糖醛、corchorifalty酸E、单棕榈酸甘油和正葵醇。结合中药系统药理学数据库和分析平台[22](traditionalChinesemedicinesystemspharmacologydatabaseandanalysisplatform,TCMSP)及文献报道,最终归纳得到了豨莶草共31种化学成分。
参考扩大范围的类药五原则,以相对分子质量<700,氢键给体数<5,氢键受体数目<10,酯水分配系数<5,以及口服生物利用度(oralbioarailability,OB)≥20%和类药性(drug-likeness,DL)>0.10作为筛选依据,分别从臭梧桐叶和豨莶草成分中筛选(并删重)得到39个化合物(表1)。采用Chem3D软件构建所有小分子的三维结构,采用MM2分子力学方法进行几何优化,优化后的结构即为分子对接的小分子初始构象。
1.2靶蛋白数据库的建立
研究表明,多种酶参与了RA的发生。非受体型酪氨酸激酶JAK1及JAK3可与炎症因子白细胞介素结合,导致RA炎症症状,因此选择性抑制JAK1及JAK3的活性治疗RA[23];受体酪氨酸激酶Syk使下游的核因子-κB(neclearfactorkappaB,NF-κB)活化,最终上调内皮细胞间黏附分子-1(intercelularadhesionmolecule-1,ICAM-1)导致RA的发生;Yeh等[24]研究表明,与RA炎症相关的转录因子,如AP-1、NF-κB及信号转导和转录活化因子的活性直接或间接地受丝裂原活化蛋白激酶(MAPK)信号转导通路的调控,而MAPK激酶1(MEK1)能够将MAPK的苏氨酸和酪氨酸残基磷酸化,使之激活,将信号转导通路上游的信号整合到MAPK,从而调节多种细胞因子的合成和促进破骨细胞分化,从而发生了RA。因此,本研究选择p38MAPK[25-26]、JAK1和JAK3[25]、Syk[26]、MEK1等5种酶建立RA靶点数据库,其3D结构从RCSBPDB数据库(https://www.rcsb.org/)下载(PDBID分别为:1OUY、4K6Z、3ZC6、4PUZ、1S9J)。采用Autodocktools1.5.6软件[27]删除晶体中多余的小分子、结晶水,为蛋白分子添加氢原子、gasteiger电荷。
1.3分子对接
1.3.1参数设置分子对接方法采用AutoDock4.2程序[28]。构象搜索采用拉马克遗传算法,群体规模:150;能量最大评估数:2500000;最大遗传代数:27000;独立运行次数:10次。其余参数均为默认设置。
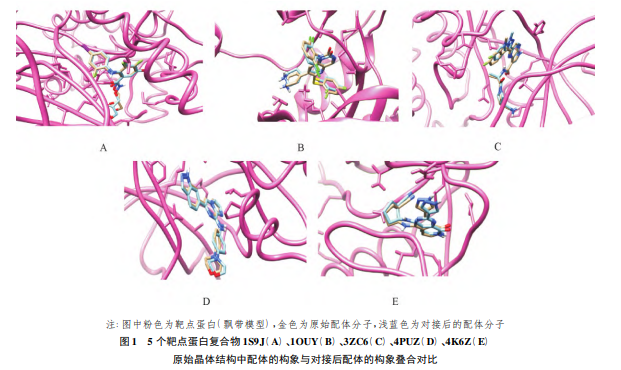
1.3.2方法可靠性验证为了验证autodock程序对本研究中对接体系的可靠性[29],分别将5种靶蛋白复合物的原有配体抽离,再重新对接到复合物的活性口袋,并计算对接后配体的构象与原始晶体结构中配体构象的均方根偏差值(root-mean-sguaredeviation,RMSD)。一般认为当RMSD值≤2.0?时,证明该对接方法可以较好地重现配体受体原来的结合模式,对接参数设置合理[30]。
1.4分子动力学模拟为了验证小分子与蛋白对接结果的合理性和稳定性,参考JacobDDurrant等[31]提出的模拟方法,对对接结合能低的化合物和相关靶蛋白的对接复合物进行了进一步的分子动力学模拟与分析。
分子动力学采用Amber18进行,大分子采用AMBERff14SB力场,小分子采用GAFF力场[32]。在HF/6-31+G(d)水平下,运用Gaussian09软件对小分子进行能量优化,并添加resp电荷。溶剂模型选择TIP3P,将蛋白置于立方体水盒子中,盒子边缘与蛋白分子距离为10?,添加抗衡离子使体系保持电中性。经过以下步骤使体系达到平衡:①限制蛋白分子,优化溶剂分子。共4000步,其中前2000步应用最陡下降法,后2000步使用共轭梯度法优化;②限制蛋白中重原子的位置的优化。共5000步,前2500步采用最陡下降法,后2500步采用共轭梯度法优化;③不加约束力整体优化。共10000步,前5000步应用最陡下降法,后5000步应用共轭梯度法;④60ps的升温过程:0~50ps内将系统温度从0K逐渐升至310K,50ps~60ps达到恒温310K;⑤50ns动力学模拟:采用NPT系统,时间步长设为2fs。
1.5网络药理学分析
1.5.1成分靶点筛选在TCMSP数据库(http://tcmspw.com/tcmsp.php)中以OB≥20%,DL>0.10为条件,筛选出豨莶草的化学成分,同时搜集成分对应的靶点信息;从Swisstargets数据库(http://www.swisstargetprediction.ch/)中检索豨桐丸4个对接结合能最低的化合物7、11、19、21(见表1)对应的靶点信息。合并删重后,将两部分靶点汇总。
1.5.2疾病靶点筛选以“RheumatoidArthritis”为关键词,在GeneCards数据库(https://www.genecards.org/)和NCBI基因数据库(https://www.ncbi.nlm.nih.gov/gene/term=)进行检索。将Genecards数据库检索到的基因根据relevance值的中位数进行2次基因筛选。
1.5.3韦恩图、蛋白质蛋白质相互作用网络构建(protein-proteininteraction,PPI)网络构建及拓扑分析筛选出的成分靶点与疾病靶点输入韦恩图作图软件Venny2.1,得到药物与疾病的交集靶点,并将其输入STRING数据库(https://string-db.org/cgi/input.pl)进行PPI网络的构建;然后将PPI网络导入Cystoscape3.6.0软件中,通过NetworkAnalyzer工具进行拓扑分析,以计算度、介度、平均最短距离和紧密度4个拓扑学参数为参考标准。通过度值排序,选取分值大于平均分的基因作为关键靶点。将关键靶点依次导入DisGeNET数据库,获取各靶点对应的蛋白类型。
1.5.4中药-成分-疾病靶点网络构建运用Cytoscape3.6.1软件构建药物-成分-疾病-靶点网络图。将1.5.1中筛选得到的靶点与中药成分进行映射关联,得到“成分-靶点”关联表,与PPI网络共同导入Cytoscape3.6.1软件,构建豨桐丸的“中药-化合物-靶点”关联网络。
1.5.5MCODE聚类分析通过MCODE分析进行RA治疗过程中关键基因的筛选。将已经构建的PPI网络导入Cytoscape3.6.1软件,通过MCODE模块进行基因簇的分析以及核心靶点的筛选。
1.5.6GO富集分析将成分与疾病的共有靶点进行GO富集分析,包括生物过程(biologicalprocess,BP)、分子功能(molecularfunction,MF)和细胞组分(cellcomponent,CC)富集。采用STRING数据库,根据P≤0.05进行筛选,使用R3.6.3软件进行柱状图绘制,选择BP、MF、CC富集程度的前10条通路作图。1.5.7KEGG通路富集将成分与疾病共有靶点进行KEGG通路富集分析,用STRING数据库,根据P值≤0.05进行筛选,采用R3.6.3软件进行气泡图的绘制。
2结果
2.1分子对接结果
2.1.1方法可靠性验证与晶体结构相比,对接后的5个复合物的RMSD值分别为:1.71、1.78、1.29、1.85、1.05?。5个靶蛋白原始晶体结构中配体构象与对接后配体的构象叠合,见图1;5个对接后的配体构象与原始晶体结构中配体构象基本重合。叠合构象和RMSD值均表明本分子对接方法和参数设置可靠。
2.1.2豨桐丸化学成分与靶蛋白对接结果以晶体原有配位作为阳性对照,将5个靶点蛋白与豨桐丸的39个成分进行对接,配体与靶蛋白的打分结果(结合能ΔG),见图2。
相关知识推荐:国家级中医期刊有哪些
对豨桐丸中39个小分子与5个靶蛋白的结合能较低的前5个化合物进行统计,共有3、7、10、11、14、17、19、21、29、38、39等11个,频率排序依次为7(5次);11、19、21(4次);38、39(3次);10、14(2次);3、17、29(1次)。因此,推测豨桐丸治疗RA可能是通过以上化合物对相关蛋白的强抑制作用来实现的。
2.1.3豆甾醇与5个靶蛋白的相互作用分析(单成分-多靶点作用)对接结果表明,化合物7(豆甾醇)与所有靶蛋白都有较低的结合能,显示其具有单成分-多靶点的作用。为了进一步分析豆甾醇和相关靶蛋白的作用机制,使用Ligplot软件[33]对豆甾醇与各靶蛋白对接的复合物的配体-受体相互作用进行分析,见图3。
2.1.44个化合物与靶蛋白3ZC6作用的模式分析(多成分-单靶点作用)化合物7、11、19和21与靶蛋白3ZC6具有较负的结合能,能有效抑制相关靶点,存在多成分-单靶点的作用机制。将化合物7、11、19、21与靶蛋白3ZC6对接的复合物的配体-受体相互作用见图3(B)和图4。
由2.1.3和2.1.4结果推测,当配体与靶蛋白结合时,非极性相互作用能够对靶蛋白和配体的结合产生促进作用,即化合物与靶蛋白结合形成较多的疏水作用时,具有较低的结合能,结合更加稳定,有效抑制靶蛋白的活性。因此,小分子与靶蛋白之间主要是通过疏水作用结合来抑制有关酶的活性,从而起到治疗RA的作用。
2.1.5豆甾醇与靶点结合的相互作用分析对接表明,豆甾醇和靶蛋白具有较强的相互作用和活性,豆甾醇由甾体骨架和一个羟基构成,为了验证其羟基是否对活性起关键作用,将其羟基替换为疏水的甲基,再次进行分子对接,结果见表2。从表2可以看出,羟基换成甲基后的化合物与5个靶蛋白的结合能均有所降低,但下降的程度不明显,表明豆甾醇与靶蛋白的主要结合力来自豆甾醇的碳骨架原子和相关氨基酸残基的疏水作用,而羟基对结合能的贡献较弱,并非关键活性基团,这为后续该药物的优化和设计提供了改造思路。
2.2分子动力学模拟结果
2.2.1分子叠合分析为了更直接的观察豆甾醇与相关靶蛋白相互作用的动力学过程,选取豆甾醇与靶蛋白4PUZ的蛋白复合物,从0ns开始,每隔5ns提取一次蛋白构象,共提取10个构象,与初始构象进行叠合,叠合结果见图5。提示,小分子虽然有构象位置的调整,但中心基本重合,表明二者结合稳定。
2.2.2RMSD分析以对接结构平衡后的构象为参照,通过分析整体蛋白结构的RMSD在模拟过程中的动态变化,进而考察复合物体系的稳定性。5个靶蛋白的骨架原子和豆甾醇的非氢原子在50ns内的RMSD变化情况,见图6。从图6(B)、图6(D)和图6(E)可看出,小分子在平衡的最初期进行了构象的调整,之后达到了稳定。图6(A)和图6(C)的小分子在25ns和30ns之间构象进行细微的调整后趋于稳定。复合物和大分子的RMSD基本稳定。RMSD值表明豆甾醇和4个靶蛋白结合稳定,实验对接结果合理可靠。
2.3网络药理学结果与分析共得到化合物7、11、19、21对应的237个靶点,豨莶草14个活性成分对应的58个靶点,将两部分靶点删重汇合共得到284个成分靶点。
2.3.1RA靶点查找经过检索,从GeneCards数据库共检索到1067个相关基因,从NCBI基因数据库共得到1206个基因。将这两个数据库的基因合并删重之后,得到与RA相关的基因共1651个。
2.3.2韦恩图将筛选出的成分靶点与疾病靶点做出韦恩图,得到112个交集靶点,作为豨桐丸治疗RA的预测靶点,如图7(A)所示。
2.3.3PPI网络构建及拓扑分析PPI网络的构建见图7(B),图中节点表示蛋白,边表示蛋白间的关联,度值越大,节点越大,在整个网络中占有的地位就越大。共筛选出42个关键靶点,将前20个靶点使用R3.6.3绘制拓扑性质如图7(C)所示。结果表明,豨桐丸治疗RA的过程中有白蛋白(ALB)、肿瘤坏死因子(TNF)、血管内皮生长因子(VEGFA)、丝裂原激活的蛋白激酶(MAPK)、SRC原癌基因(SRC)、半胱天冬酶(CASP3)、前列腺素G/H合成酶2(PTGS2)、基质金属蛋白酶(MMP9)等多种物质。——论文作者:庄严,王少佩,王永林,胡玉龙,仝艳,董春红,李晓飞


